クリプトクロム光受容タンパク質による光応答シグナル伝達の中間体構造を解明
2025年5月17日
大阪大学
東北大学
高輝度光科学研究センター
【研究成果のポイント】
◆青色光受容タンパク質であるクリプトクロムが光を検知した後の中間体構造を経時的に解析することで、クリプトクロムの光応答機構の詳細を解明
◆クリプトクロムに内包されるフラビン補酵素(FAD)の光還元反応に伴い、FAD近傍とFAD遠位にて独立に構造変化が起こることで、シグナル伝達を担う分子構造へと遷移することを発見
◆クリプトクロムの構造-機能相関の理解が深まり、今後の人工光遺伝学ツール開発の発展に貢献
|
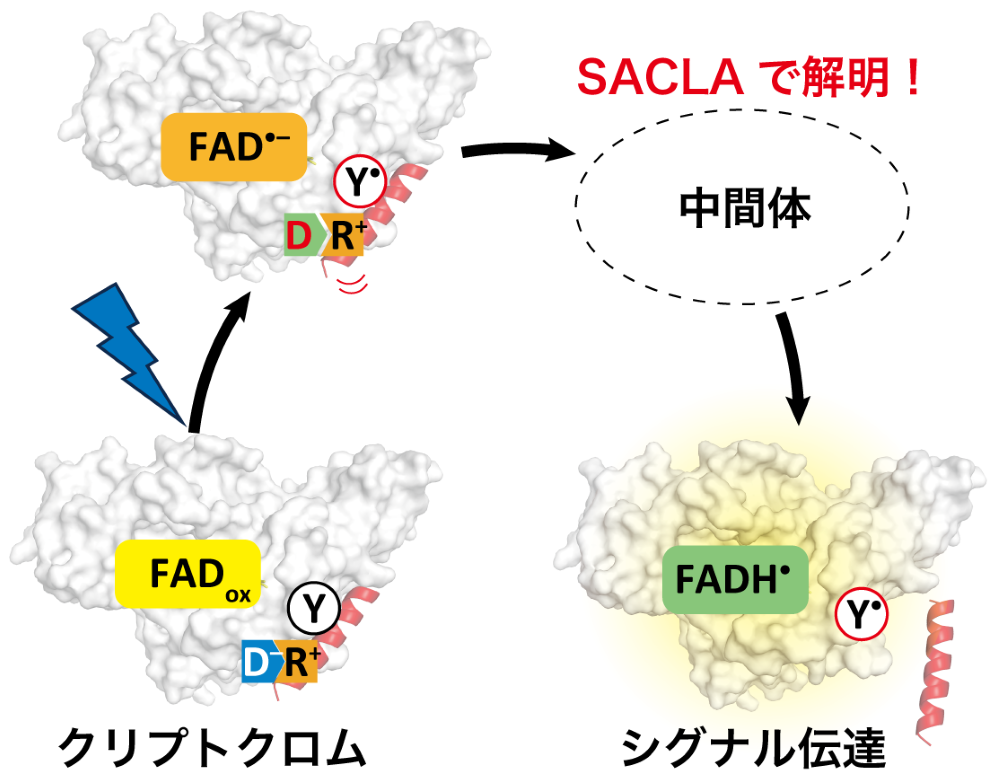
台湾大学のManuel Maestre-Reyna助理教授、ドイツ・フィリップ大マールブルグのLars-Oliver Essen教授、大阪大学大学院基礎工学研究科の山元淳平准教授、台湾中央研究院・生物化學研究所の蔡明道特聘研究員らは、理化学研究所の別所義隆客員研究員、公益財団法人高輝度光科学研究センターの大和田成起主幹研究員、東北大学の南後恵理子教授、京都大学の岩田想教授、兵庫県立大学の當舎武彦教授、名古屋大学の梅名泰史准教授、およびグルノーブル・アルプ大学、欧州シンクロトロン放射光研究所の研究者らとの国際共同研究にて、緑藻類をはじめとした植物やハエの内に存在する青色光受容タンパク質であるクリプトクロムの、光受容後10ナノ秒から233ミリ秒にわたる中間体の立体構造を解明しました(図1)。
図1 青色光受容クリプトクロムの光応答反応の概要
【山元准教授のコメント】
|
研究の背景
ギリシャ語で「隠れた色素」を意味するクリプトクロムは、植物の生育やシグナル伝達・概日リズム形成などに関与する多機能タンパク質です。その内、青色光に応答して機能するものはフラビンアデニンジヌクレオチド(FAD)をタンパク質内部に有しています。タンパク質中のFADが光によって励起されると、FAD近傍に存在する芳香族アミノ酸側鎖から電子を獲得し、一電子還元状態のアニオン型FADラジカル(FAD•–)と一電子酸化状態のアミノ酸側鎖ラジカルを1ナノ秒以内に生成します。
この光還元反応によって生成するラジカル対はクリプトクロムの光応答機能に関与することが知られていましたが、光還元反応がどのように機能発現につながるのかは不明でした。
研究の内容
今回、研究グループは、青色光受容クリプトクロムの一つであるクラミドモナス由来動物型クリプトクロム(CraCRY)の微結晶をターゲットとし、まずは異なるFAD酸化状態を有するCraCRYの三次元構造をSACLAにて明らかにしました。酸化状態(FADox)および二電子還元状態FAD(FADH–)を有するCraCRYでは全体構造は概ね一致しました。一方で、一電子還元状態中性FADラジカル(FADH•)を有するCraCRYはC末端領域において電子密度の喪失が認められ、この部分の構造を決定することができませんでした。このことから、FADoxからFADH•への変化に伴うタンパク質構造変化が光応答機能発現の鍵となることを見出しました。
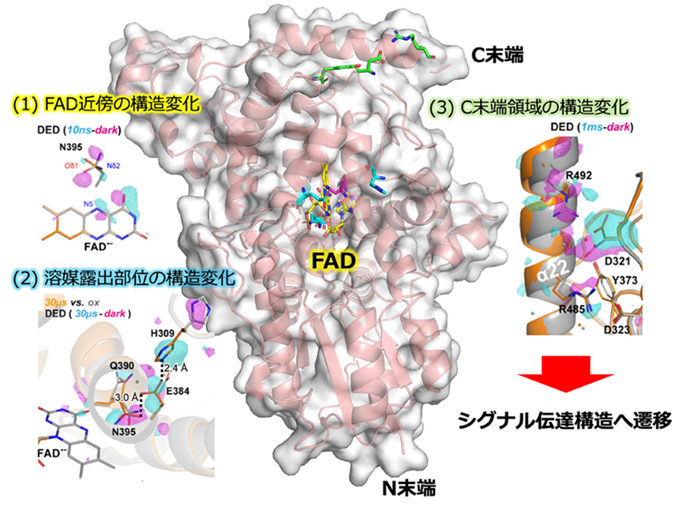
続いて、FADoxを有するCraCRY微結晶を用いて、光励起後10ナノ秒から233ミリ秒における反応中間体の三次元構造をTR-SFXによって解明しました。その結果、(i) FAD近傍、(ii) FADに隣接する溶媒露出部位、および(iii) C末端領域の3箇所が、異なる時間領域で構造変化することがわかりました(図2)。まず、FAD•–の形成に応じてFAD近傍が構造変化し、その後マイクロ秒以降に溶媒露出部位において逐次的に構造変化が起こることで、FAD•–のプロトン化産物であるFADH•の形成を時空間的に制御することを見出しました。また、FAD光還元反応によって生じる一電子酸化状態のアミノ酸側鎖ラジカルの近傍にはC末端領域が存在し、ラジカルの形成によってC末端領域とタンパク質本体をつなぐ塩橋が崩壊することで、C末端領域の構造変化が独立して誘起されることを明らかにしました。これらのデータから、クリプトクロムが光に応答して下流因子へとシグナル伝達を担う構造へと遷移する分子機構を解明しました。
図2 光受容後の構造変化の概要
本研究成果が社会に与える影響(本研究成果の意義)
本研究では、青色光受容クリプトクロムが光を検知した後の構造を経時的に解析することで、クリプトクロムの光応答機構の詳細な描像を与えることができたことから、基礎科学の理解に大きく貢献します。
また、クリプトクロムはオプトジェネティクス(光遺伝学)のツールの一つとなりうることが示唆されており、今回の研究から、より高活性な人工光遺伝学ツールの創成などへの応用研究への道が開けました。
特記事項
なお、本研究は、主に日本学術振興会 科学研究費助成事業新学術領域研究(研究領域提案型)「二元機能性青色光受容タンパク質の光応答機構」(代表者:山元淳平)、同基盤研究(C)「DNA光回復酵素フォトリアーゼのXFEL時分割結晶構造解析」(代表者:別所義隆)、JST創発的研究推進事業(FOREST)「DNA修復反応の動的構造解析基盤の創出」(代表者:山元淳平)、日本医療研究開発機構(AMED)の生命科学・創薬研究支援基盤事業(BINDS)(代表者:岩田想、分担者:南後恵理子)による助成を受けて行われました。
【用語解説】
※1. フラビンアデニンジヌクレオチド
ビタミンB2(リボフラビン)にピロリン酸を介してアデノシンが結合した化合物で、多くの酸化還元酵素の補酵素として使われている。アデノシンが結合しないフラビンモノヌクレオチド(FMN)とともに、フラビン補酵素と呼ばれる。酸化型、一電子還元型アニオン型ラジカル、一電子還元型中性ラジカル、二電子還元型の酸化還元状態をとり、それぞれFADox、FAD•–、FADH•、FADH–と示した。
※2. 光還元反応
クリプトクロムが属する光回復酵素・クリプトクロムスーパーファミリー(PCSf)に特徴的な光依存的なFADの還元反応。励起状態のFAD発色団は、PCSf中にて高度に保存された3つないし4つの芳香族アミノ酸側鎖から電子を獲得し、還元状態のFAD種が生成する。一方で、芳香族アミノ酸側鎖上に生じた正孔は、保存された他のアミノ酸側鎖から連続的に電子授受が起こることでタンパク質外縁近傍へと移動し、還元状態のFAD種が安定化される。
※3. X線自由電子レーザー(XFEL)
近年の加速器技術の発展によって実現したX線領域のパルスレーザー。従来の半導体や気体を発振媒体とするレーザーとは異なり、真空中を高速で移動する電子ビームを媒体とするため、原理的な波長の制限はない。「SPring-8(スプリングエイト)」などの従来の放射光源と比較して、10億倍も高い輝度のX線がフェムト秒(1,000兆分の1秒)スケールの時間幅を持つパルス光として出射される。この高い輝度を活かしてマイクロメートルサイズの小さな結晶を用いたタンパク質の原子分解能の構造解析やX線領域の非線形光学現象の解明などの用途に用いられている。
XFELはX-ray Free Electron Laserの略。
※4. SACLA
理化学研究所と高輝度光科学研究センターが共同で建設した日本ではじめてのXFEL施設。高い空間コヒーレンス、短いパルス幅、高いピーク輝度を備えたX線領域のレーザーを発生させる。2011年3月に施設が完成し、SPring-8 Angstrom Compact free electron LAserの頭の頭文字を取ってSACLAと命名された。2011年6月に最初のX線レーザーを発振、2012年3月から共用運転が開始され、利用実験が始まった。大きさが諸外国の同様の施設と比べて数分の1とコンパクトであるにもかかわらず、0.1 nm以下という世界最短クラスの波長のレーザー生成能力を持つ。
※5. 時分割シリアルフェムト秒X線結晶構造解析(TR-SFX)
結晶中の分子の微細な動きを高い時間・空間分解能で観察する手法。本研究では、高粘度媒体に懸濁させた微結晶をXFELおよび励起パルスレーザー光の焦点に対して連続的に吐出することで、光励起後一定の遅延時間における回折像を取得した。数万枚のイメージデータからタンパク質の立体構造を決定し、光応答反応中間体のスナップショットを構築した。
|
本件に関するお問い合わせ先 |
本件に関するお問い合わせ先
<研究に関するお問い合わせ>
大阪大学 大学院基礎工学研究科 准教授
山元淳平(やまもと じゅんぺい)
東北大学 多元物質科学研究所 教授
南後恵理子(なんご えりこ)
公益財団法人高輝度光科学研究センター 主幹研究員
大和田成起(おおわだ しげき)
<広報に関するお問い合わせ>
大阪大学 基礎工学研究科 庶務係
TEL: 06-6850-6131 FAX: 06-6850-6477
E-mail: ki-syomu office.osaka-u.ac.jp
office.osaka-u.ac.jp
東北大学 多元物質科学研究所 広報情報室
TEL: 022-217-5198
E-mail: press.tagen grp.tohoku.ac.jp
grp.tohoku.ac.jp
公益財団法人高輝度光科学研究センター
利用推進部 普及情報課
TEL: 0791-58-2785
E-mail: このメールアドレスはスパムボットから保護されています。閲覧するにはJavaScriptを有効にする必要があります。
(SPring-8 / SACLAに関すること)
公益財団法人高輝度光科学研究センター
利用推進部 普及情報課
TEL:0791-58-2785 FAX:0791-58-2786
E-mail:このメールアドレスはスパムボットから保護されています。閲覧するにはJavaScriptを有効にする必要があります。